Недавно я обнаружил весьма печальный факт: на Хабре совершенно не освещена такая забавная тема, как ДНК-оригами. Есть только один пост 2009 года, рассказывающий лишь самое начало занимательной истории о том, как из ДНК (да-да, той самой дезоксирибонуклеиновой кислоты, несущей нашу генетическую информацию) можно создавать всякие хитрые, плоские и трехмерные штуки нанометрового размера. Та самая нано-технология, как она есть. В этом обзоре я хочу рассказать о развитии ДНК-оригами: двухмерные смайлики из ДНК, трехмерные фигуры, кристаллы из ДНК с запрограммированной структурой, ДНК-«коробочки» с крышкой, способные нести молекулы нужных веществ и выпускать их после сигнала об открытии крышки, и, наконец, динамические структуры типа ДНК-шагохода (walker), гуляющего по подложке (создатели гордо говорят, что это уже наноробот!). Кто хочет узнать больше о том, зачем все это нужно, почитать о технологиях изготовления красивых нанометровых штук из ДНК или просто посмотреть красивые картинки, добро пожаловать под кат.
Так выглядит ДНК-наноробот
В конце двадцатого — начале двадцать первого века встал вопрос о конструировании объектов нанометрового размера. Для чего? Общий вектор на миниатюризацию существует достаточно давно, причем исторически это всегда было движение «сверху вниз» — например, в 70-х годах при изготовлении микросхем минимальный контролируемый размер составлял 2-8 мкм, далее это значение стремительно уменьшалось и сейчас в серийном производстве находятся чипы, выполненные по 22-нм технологическому процессу. Тут у думающих людей возник вопрос: а нельзя ли двигаться «снизу вверх»? Нельзя ли заставить атомы и молекулы собираться в нужные структуры и затем эти структуры использовать в технике? Очевидны требования к такой «самособирающейся» системе: материалы для нее должны быть достаточно дешевыми и доступными, самосборка сложной пространственной структуры системы должна легко и очевидно «программироваться», система должна быть способна нести полезный функционал. Тут же вспомнили, что в природе такие самособирающиеся системы уже существуют и прекрасно работают — это макромолекулы всех живых организмов, например, белки. Здесь приходит и первое разочаровние — белки слишком сложно устроены, их трехмерная структура задается совершенно неочевидным образом множеством нековалентных взаимодействий и получить белок с произвольной структурой — до сих пор абсолютно нетривиальная и нерешаемая задача. То есть использовать белки для конструирования нужных объектов нано-размеров технически невозможно. Что же делать? Оказывается, есть и другие макромолекулы, чья структура устроена гораздо проще структуры белков.
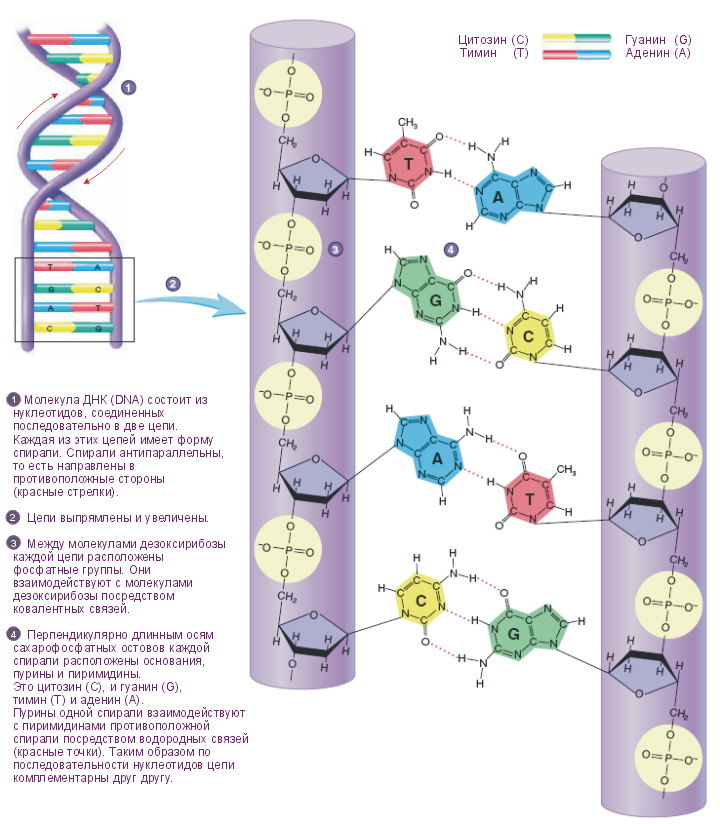
 В 1953 году Уотсон и Крик опубликовали свою модель структуры ДНК, оказавшейся абсолютно верной. ДНК (дезоксирибонуклеиновая кислота) — это интересно устроенный линейный полимер. Одна нить ДНК состоит из монотонно повторяющегося сахаро-фосфатного остова (он асимметричен и имеет направление, различают 5' и 3' конец цепи), однако к каждому сахару (дезоксирибозе в случае ДНК) прикреплен один из четырех нуклеотидов (синоним слова нуклеотид — «основание») — аденин, либо тимин, либо цитозин, либо гуанин. Обычно их обозначают одной буквой — А, Т, Ц, Г. Таким образом, в ДНК есть только 4 типа мономеров, в отличие от 20 аминокислот в составе белка, что делает структуру ДНК намного проще. Дальше становится еще веселей — есть так называемое «Уотсон-Криковское спаривание оснований»: аденин может специфично связываться с тимином, а гуанин — с цитозином, образуя пары А-Т и Г-Ц (и еще Т-А и Ц-Г, разумеется), другие взаимодействий между нуклеотидами в упрощенном случае можно считать невозможными (они возможны в виде исключения при некоторых редких условиях, но для нас это не важно). Уотсон-Криковское спаривание оснований еще называется комплементарностью.
В 1953 году Уотсон и Крик опубликовали свою модель структуры ДНК, оказавшейся абсолютно верной. ДНК (дезоксирибонуклеиновая кислота) — это интересно устроенный линейный полимер. Одна нить ДНК состоит из монотонно повторяющегося сахаро-фосфатного остова (он асимметричен и имеет направление, различают 5' и 3' конец цепи), однако к каждому сахару (дезоксирибозе в случае ДНК) прикреплен один из четырех нуклеотидов (синоним слова нуклеотид — «основание») — аденин, либо тимин, либо цитозин, либо гуанин. Обычно их обозначают одной буквой — А, Т, Ц, Г. Таким образом, в ДНК есть только 4 типа мономеров, в отличие от 20 аминокислот в составе белка, что делает структуру ДНК намного проще. Дальше становится еще веселей — есть так называемое «Уотсон-Криковское спаривание оснований»: аденин может специфично связываться с тимином, а гуанин — с цитозином, образуя пары А-Т и Г-Ц (и еще Т-А и Ц-Г, разумеется), другие взаимодействий между нуклеотидами в упрощенном случае можно считать невозможными (они возможны в виде исключения при некоторых редких условиях, но для нас это не важно). Уотсон-Криковское спаривание оснований еще называется комплементарностью.
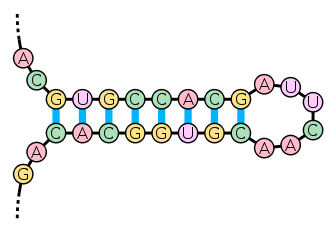
Две цепи ДНК, последовательность оснований которых комплементарна, немедленно «слипаются» в двойную спираль.Возникает вопрос: а что, если на одной цепи ДНК находятся две комплементарные области? Ответ: цепь ДНК может согнуться и комплементарные области смогут образовать двойную спираль, а вместе с местом изгиба эта структура будет называться «шпилькой» (DNA hairpin):
На чем же основано «слипание» двух комплементарных цепей ДНК (или, аналогично, двух комплементарных участков одной цепи)? Это взаимодействие держится на водородных связях. Пара А-Т соединяется двумя водородными связями, пара Г-Ц — тремя, поэтому эта пара более энергетически устойчива. Про водородные связи надо понимать следующее: энергия одной водородной связи (5 ккал/моль) не намного превосходит энергию теплового движения, а значит, одна отдельно взятая водородная связь может быть с высокой вероятностью тепловым движением разрушена. Однако, чем больше водородных связей, тем более устойчивой становится система. Это значит, что короткие участки комплементарных оснований ДНК не могут образовать устойчивую двойную спираль, она будет легко «плавиться», однако более длинные комплементарные участки уже смогут образовать стабильные структуры. Стабильность двухцепочечной структуры выражается одним параметром — температурой плавления (Тм, melting temperature). По определению, температура плавления — это температура, при которой в равновесии 50% молекул ДНК с данной длиной и последовательностью нуклеотидов находятся в двухцепочечном состоянии, а другие 50% — в расплавленном одноцепочечном состоянии. Очевидно, что температура плавления напрямую зависит от длины комплементарной области (чем длиннее — тем выше температура плавления) и от нуклеотидного состава (так как в паре Г-Ц три водородные связи, а в паре А-Т — две, то чем больше пар Г-Ц, тем выше температура плавления). Температура плавления для данной последовательности ДНК легко считается по эмпирически выведенной формуле.
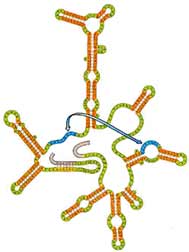
Итак, теорию мы изучили. Что же мы можем сделать на практике? С помощью химического синтеза мы можем напрямую синтезировать цепи ДНК длиной до 120 нуклеотидов (просто потом выход продукта резко падает). Если же нам нужна более длинная цепь, то ее без проблем можно собрать из тех самых химически синтезированных фрагментов длиной до 120 нуклеотидов (например, дядюшка Крейг Вентер отличился тем, что из кусочков собрал ДНК длиной аж 1,08 миллиона пар оснований). То есть в 21 веке мы можем легко и дешево делать ДНК любой последовательности, какой только захотим. А хотим мы, чтобы потом ДНК сворачивалась во всякие хитрые и сложные структуры, которые мы потом сможем использовать. Для этого у нас есть принцип комплементарности — как только в последовательности ДНК появляются комплементарные зоны, они слипаются и образуют двухцепочечный участок. Очевидно, мы хотим делать структуры, стабильные при комнатной температуре, значит мы хотим рассчитать температуру плавления для данных участков и сделать ее достаточно большой. При этом на одной цепи ДНК мы можем делать много разных областей с разными последовательностями и слипаться будут только комплементарные. Так как комплементарных областей может быть несколько, в результате молекула может свернуться достаточно сложным образом! Как-то так, например:
Так же, помимо положительного дизайна (создание областей, способных образовывать нужную нам структуру), при разработке структур с самосборкой нельзя забывать и о негативном дизайне — нужно проверять получившуюся последовательность ДНК на потенциальное наличие паразитных взаимодействий (когда части созданных нами областей оказываются способными взаимодейстовать по-другому, образуя ненужные нам паразитные структуры) и от этих паразитных структур и взаимодействий избавляться, меняя нуклеотидную последовательность ДНК. Как получить простейшие структуры ДНК типа «шпильки» достаточно очевидно, но скучно и неинтересно. Можно ли из ДНК сделать что-то посложнее? Здесь уже без компьютерных вычислений не обойтись. Мы хотим некую структуру и теперь должны подобрать последовательность ДНК, которая в эту структуру свернется за счет взаимодействия комплементарных областей, но при этом в последовательности не должно быть паразитных взаимодействий, непредусмотренных нами комплементарных областей, образующих альтернативные структуры. Плюс структура должна отвечать другим критериям, например, иметь температуру плавления выше некоторой заданной величины. В результате имеем типичную оптимизационную задачу.
Методологический прорыв устроил Paul Rothemund (Калифорнийский Технологический Институт) в 2006 году, именно он и придумал термин «ДНК-оригами». В своей статье в «Nature» он представил множество забавных двухмерных объектов, сделанных из ДНК. Принцип, предложенный им, достаточно прост: взять длинную (примерно 7000 нуклеотидов )«опорную» одноцепочечную молекулу ДНК и затем с помощью сотни коротких ДНК-скрепок, образующих двухцепочечные области с опорной молекулой, согнуть опорную ДНК в нужную нам двухмерную структуру. Вот рисунок из оригинальной статьи, представляющий все стадии разработки. Для начала (а) нарисуем нужную нам форму красным цветом и прикинем, как заполнить ее ДНК (представим ее на этом этапе в виде труб). Далее (b) представим, как провести одну длинную опорную молекулу по нужной нам форме (показана черной линией). На третьем этапе (с) подумаем, где мы хотим разместить «скрепки», стабилизирующие укладку длинной опорной цепи. Четвертый этап (d): больше деталей, прикидываем, как будет выглядеть вся нужная нам структура ДНК и, наконец, (e) мы имеем схему нужной нам структуры, можно заказывать ДНК нужной последовательности!
Как же из химически синтезированных ДНК собрать нужную нам структуру? Здесь на помощь приходит процесс плавления. Мы берем пробирку с водным раствором, бросаем в нее все фрагменты ДНК и нагреваем до 94-98С, температуры, которая гарантировано плавит всю ДНК (переводит ее в одноцепочечную форму). Далее мы просто очень медленно (в течении многих часов, в некоторых работах — в течении нескольких дней) охлаждаем пробирку до комнатной температуры (эта процедура называется «отжиг», annealing). При этом медленном охлаждении, когда температура оказывается достаточно низкой, постепенно образуются нужные нам двухцепочечные структуры. В оригинальной работе в каждом эксперименте примерно 70% молекул успешно собирались в нужную структуру, остальные имели дефекты.
Далее, после того, как структура рассчитана, неплохо бы доказать, что она собирается именно так, как нам надо. Для этого чаще всего используют атомно-силовую микроскопию, которая как раз прекрасно показывает общую форму молекул, но иногда используют и cryo-EM (электронную микроскопию). Автор сделал множество веселых форм из ДНК, на картинках представлены расчетные структуры и результат экспериментального определения структур с помощью атомно-силовой микроскопии. Наслаждайтесь!


После того, как разобрались с конструированием сложных плоских объектов, почему бы не перейти к третьему измерению? Здесь пионерами была группа ребят из Института Скриппса в Ла-Холле, Калифорния, которые в 2004 году придумали, как из ДНК сделать нано-октаэдр. Хотя эта работа и сделана на 2 года раньше плоского ДНК-оригами, в тот раз был решен лишь частный случай (получение октаэдра из ДНК), а в работе по ДНК-оригами было предложено общее решение, поэтому именно работа 2006 года по ДНК-оригами считается основополагающей.
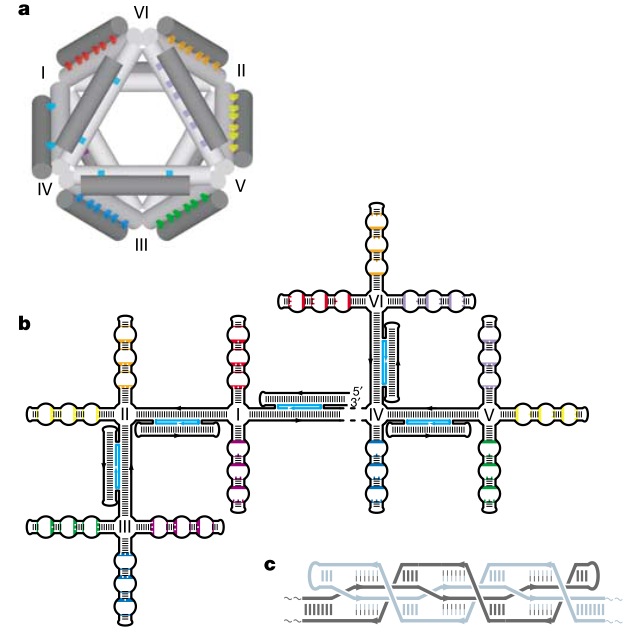
Октаэдр был сделан из одноцепочечной молекулы ДНК длиной примерно 1700 нуклеотидов, имеющей комплементарные области и к тому же скрепленой пятью 40-нуклеотидными ДНК-адаптерами, в результате был получен октаэдр с диаметром 22 нанометра.
На рисунке обратите внимание на цветовую кодировку на двухмерной развертке октаэдра. Видите области, отмеченные одинаковым цветом? Они содержат как комплементарные зоны (параллельные участки соединенные поперечными связями), так и некомплементарные (на схеме они изображены в виде пузырьков), при этом зоны одного цвета, расположенные в разных частях двухмерной развертки, взаимодействуют друг с другом, образуя сложную структуру, изображенную на рисунке 1с и образующую грань трехмерного тетраэдра. Наслаждайтесь красивыми картинками!

В 2009 году ученые из Бостона и Гарвардского Университета опубликовали принципы построения трехмерного ДНК-оригами, как они сами говорят, по подобию пчелиных сот. Одно из достижений этой работы — люди написали open-source программу caDNAno для конструирования трехмерных структур ДНК (она работает на Autodesk Maya). С этой программой даже неспециалист может собрать нужную структуру из готовых блоков с использованием простенького графического интерфейса, а программа рассчитает необходимую последовательность (или последовательности) ДНК, в эту структуру сворачивающуюся.

В следующей своей работе они научились делать из ДНК сложные трехмерные объекты с контролируемым искривлением и порадовали читателей журнала «Science» красивыми картинками разных искривленных объектов из ДНК (там такие классные шестеренки получились!).

 До сих пор ученые игрались с непериодическими структурами из ДНК. А что если сделать такую структуру, чтобы один блок мог взаимодействовать с другим таким же блоком, и так до бесконечности? Представьте себе три отрезка, расположенных под углом 90 градусов друг к другу (походит на противотанковый еж). Очевидно, что такая структура может быть узлом бесконечной кубической решетки, если каждая сторона такого ежа будет взаимодействовать с другим таким ежом. Именно эту идею в 2010 году воплотили на практике ученые из Нью-Йорка, они сделали такого ДНК-ежа, который немедленно сформировал трехмерную решетку, то есть кристалл из ДНК, так что они использовали рентгеноструктурный анализ, чтобы показать, что ДНК образовали именно такую структуру, какую они и хотели. В свою очередь, так как кристаллы ДНК имели размер до пол-миллиметра (а это уже макро-объект), было гордо заявлено, что теперь из нано-объектов мы умеем собирать макро-объекты.
До сих пор ученые игрались с непериодическими структурами из ДНК. А что если сделать такую структуру, чтобы один блок мог взаимодействовать с другим таким же блоком, и так до бесконечности? Представьте себе три отрезка, расположенных под углом 90 градусов друг к другу (походит на противотанковый еж). Очевидно, что такая структура может быть узлом бесконечной кубической решетки, если каждая сторона такого ежа будет взаимодействовать с другим таким ежом. Именно эту идею в 2010 году воплотили на практике ученые из Нью-Йорка, они сделали такого ДНК-ежа, который немедленно сформировал трехмерную решетку, то есть кристалл из ДНК, так что они использовали рентгеноструктурный анализ, чтобы показать, что ДНК образовали именно такую структуру, какую они и хотели. В свою очередь, так как кристаллы ДНК имели размер до пол-миллиметра (а это уже макро-объект), было гордо заявлено, что теперь из нано-объектов мы умеем собирать макро-объекты.
Вот стерео-картинка узла решетки, если умеете правильно скашивать глаза (это прямая стереопара), можете посмотреть в 3D (на нижней картинке с электронной плотностью ДНК четко видны два узла-«противотанковых ежа»):

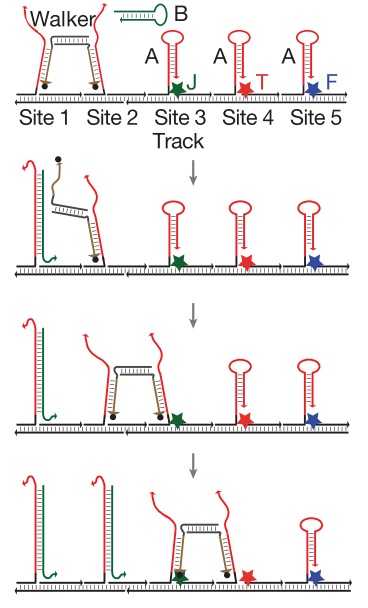
Следующий шаг в конструировании трехмерных нано-объектов так же очевиден — а что если заставить это все как-то двигаться? Движущийся объект уже можно и нано-роботом назвать. Самый простенький ДНК-шагоход был сделан в 2008 году командой из Калифорнийского Технологического института. Работает он по достаточно простому принципу. Представьте себе одноцепочечную ДНК длиной, скажем, 100 нуклеотидов. Представьте другую одноцепочечную ДНК, короткую, длиной 50 нуклеотидов, комплементарную половине первой молекулы. Что будет, если их смешать? Правильно, они образуют двухцепочечную структуру в районе этих 50 нуклеотидов, вторая же половина первой молекулы останется свободной. А что если к этой структуре добавить еще одну молекулу ДНК, длиной 100 нуклеотидов и полностью комплементарной первой молекуле? Ответ вполне очевиден: она вытеснит короткую цепь длиной 50 нуклеотидов, так как обладает большим сродством к первой молекуле (у них сродство 100 из 100, а с короткой — только 50 из 100 нуклеотидов). Именно так и работал первый ДНК-шагоход. На подложке закреплены молекулы одноцепочечной ДНК, к ним из раствора приходит молекула-шагоход, имеющая комплементарные зоны к двум соседним цепям на подложке и связывается с ними. Если потом мы добавим в раствор другую ДНК, имеющую большее сродство к первой цепи на подложке, то она вытеснит одну ногу шагохода, после чего эта нога свяжется со следующей (третьей) цепью ДНК на подложке. Добавляя новые вытесняющие ДНК можно гнать шагоход все дальше и дальше по подложке. Обратный ход невозможен, так как предыдущие цепи на подложке уже инактивированы связыванием с более длинной молекулой ДНК.

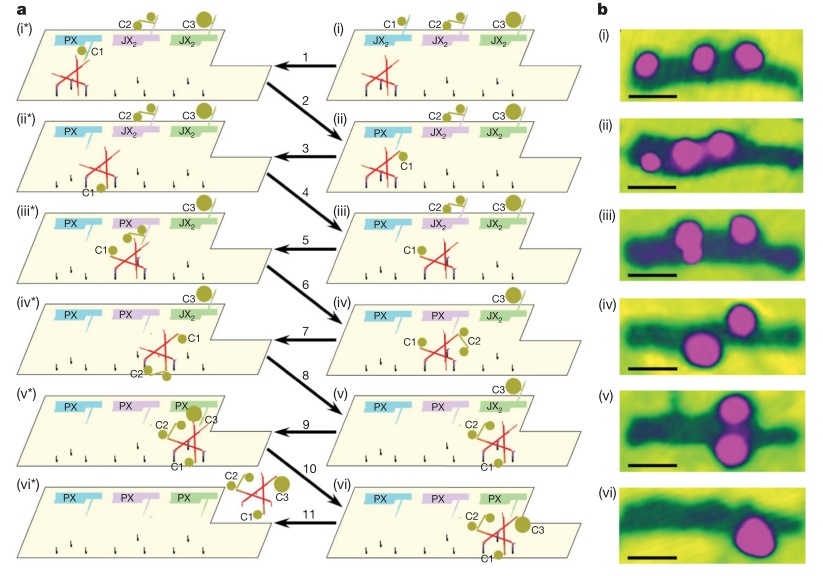
Хотя первый шагоход выглядел настолько примитивно, конструкция была доработана учеными из Нью-Йорка. Они сделали более сложный шагоход с несколькими «руками» и «ногами», прицепили при помощи комплементарных ДНК на подложку «груз» (золотые частицы диаметром 5 и 10 нм) и «запрограммировали» шагоход таким образом, чтобы он прошел по подложке и собрал груз — три маленькие и одну большую золотые частицы. Последовательность шагов легко отследить по стрелочкам, а на экспериментальной картинке справа видны частицы золота и как шагоход их собирает. Нано-робот в действии! На нижней картинке показано, как именно происходит процесс «шагания» и сбора груза, принцип — тот же самый, вытеснение одной ДНК другой.
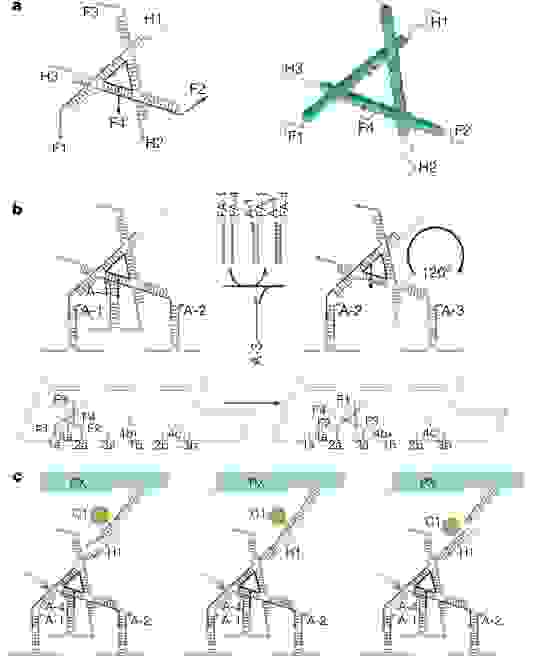
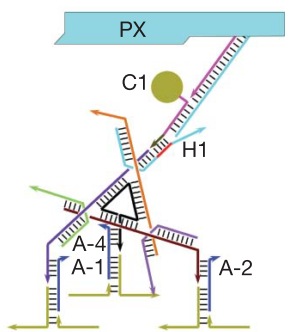
Но это еще не все. Венцом ДНК-робототехники (думаю, тут в моем голосе слышим некоторый сарказм) стал «наноробот для молекулярного транспорта», как окрестили его создатели из Бостона. Фактически ребята сделали некую «коробочку» из ДНК, закрывающуюся на «замок» из ДНК, который может быть открыт по уже известному нам принципу вытеснения одной ДНК другой, как в шагоходах. Внутри коробочки спрятан груз — частицы золота либо молекулы иммуноглобулина. ДНК можно химически модифицировать таким образом, чтобы на ней можно было закрепить этот самый груз. Итак, мы имеем закрытую коробочку, содержимое коробочки надежно спрятано. Тут мы добавляем молекулу, открывающую коробочку, она открывается и иммуноглобулин, спрятанный внутри, выходит наружу и начинает действовать! Мы же хлопаем в ладоши, умиляемся и радуемся прогрессу.
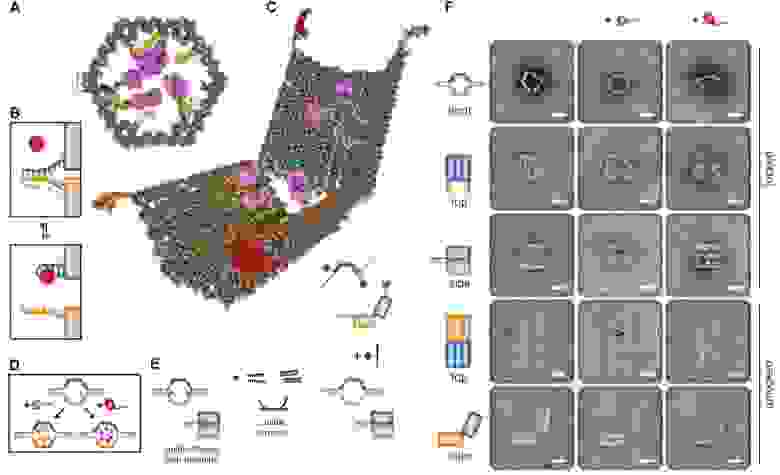
Ребята даже не поленились сделать демонстрацию proof of principle на живых раковых клетках: они прятали в коробочку антитела, блокирующие ключевые белки клеточного цикла, вводили коробочки в раковые клетки и после добавления открывающего коробочку активатора раковые клетки правда переставали делиться! Таким образом была показана принципиальная возможность использования таких конструкций для направленной доставки лекарств в организме и их выделения в нужное время по сигналу от молекулы-активатора. Одна осталась проблема, как раковую клетку от здоровой надежно отличить…
Все это, конечно, здорово и хорошо, картинки красивые, но внимательный читатель может спросить: «А где обещанная в самом начале польза для народного хозяйства?». Как и со всякой новой технологией, пока большой практической пользы действительно нет, помимо эстетического удовольствия от созерцания этой нано-красоты. Однако, все только начинается! Во-первых, ДНК может быть химически модифицирована, к ней могут быть добавлены химические группы, обеспечивающие связывание с другими молекулами и тогда ДНК можно использовать как подложку для построения сложных структур из других молекул. Например, сейчас все хотят что-то мастерить из нанотрубок. Если удастся сделать адаптер, связывающийся одним концом с ДНК, а другим — с нанотрубкой, тогда структуры из ДНК можно использовать для соединения нанотрубок. С другой стороны, уже есть сообщения о контролируемой металлизации ДНК, а отсюда уже рукой подать до конструирования электронных устройств на базе структур из металлизированной ДНК. Может быть, ученые изобретут более подходящий полимер, обеспечивающий более удобную самосборку сложных структур, но в любом случае ДНК-оригами займет свое место в истории науки, как один из первых примеров конструирования сложных объектов в нано-масштабе. Как бы то ни было, нас ждет большое и светлое будущее, чего и вам желаю!
PS: интересное дополнение от vxsw:
«Уже пару лет проводится конкурс BIOMOD по дизайну таких штук при поддержке Wyss Institute (замеченного во многих интересных биотехнологических достижениях вроде недавней 3D-печати мини-аккумуляторов).»
PPS: в личке спросили, почему ДНК, а не РНК. Ответ такой: я вижу две основные причины: (1) ДНК — химически более стабильна. Все живые организмы синтезируют огромное количества РНКаз, ферментов, уничтожающих РНК. Если Вы случайно залезете голым пальцем в пробирку с РНК, от РНК ничего не останется — все сожрут РНКазы. Поэтому с РНК работают в специальных помещениях и тд — мороки гораздо больше, чем при работе с ДНК. С ДНК таких проблем нет, палец в пробирку сунешь — ничего ДНК не будет. (2) Стоимость химического синтеза РНК в разы превышает стоимость синтеза ДНК. Думаю, поэтому народ и развлекается с ДНК — дешевле и проще.
Так выглядит ДНК-наноробот
Немного теории
В конце двадцатого — начале двадцать первого века встал вопрос о конструировании объектов нанометрового размера. Для чего? Общий вектор на миниатюризацию существует достаточно давно, причем исторически это всегда было движение «сверху вниз» — например, в 70-х годах при изготовлении микросхем минимальный контролируемый размер составлял 2-8 мкм, далее это значение стремительно уменьшалось и сейчас в серийном производстве находятся чипы, выполненные по 22-нм технологическому процессу. Тут у думающих людей возник вопрос: а нельзя ли двигаться «снизу вверх»? Нельзя ли заставить атомы и молекулы собираться в нужные структуры и затем эти структуры использовать в технике? Очевидны требования к такой «самособирающейся» системе: материалы для нее должны быть достаточно дешевыми и доступными, самосборка сложной пространственной структуры системы должна легко и очевидно «программироваться», система должна быть способна нести полезный функционал. Тут же вспомнили, что в природе такие самособирающиеся системы уже существуют и прекрасно работают — это макромолекулы всех живых организмов, например, белки. Здесь приходит и первое разочаровние — белки слишком сложно устроены, их трехмерная структура задается совершенно неочевидным образом множеством нековалентных взаимодействий и получить белок с произвольной структурой — до сих пор абсолютно нетривиальная и нерешаемая задача. То есть использовать белки для конструирования нужных объектов нано-размеров технически невозможно. Что же делать? Оказывается, есть и другие макромолекулы, чья структура устроена гораздо проще структуры белков.
 В 1953 году Уотсон и Крик опубликовали свою модель структуры ДНК, оказавшейся абсолютно верной. ДНК (дезоксирибонуклеиновая кислота) — это интересно устроенный линейный полимер. Одна нить ДНК состоит из монотонно повторяющегося сахаро-фосфатного остова (он асимметричен и имеет направление, различают 5' и 3' конец цепи), однако к каждому сахару (дезоксирибозе в случае ДНК) прикреплен один из четырех нуклеотидов (синоним слова нуклеотид — «основание») — аденин, либо тимин, либо цитозин, либо гуанин. Обычно их обозначают одной буквой — А, Т, Ц, Г. Таким образом, в ДНК есть только 4 типа мономеров, в отличие от 20 аминокислот в составе белка, что делает структуру ДНК намного проще. Дальше становится еще веселей — есть так называемое «Уотсон-Криковское спаривание оснований»: аденин может специфично связываться с тимином, а гуанин — с цитозином, образуя пары А-Т и Г-Ц (и еще Т-А и Ц-Г, разумеется), другие взаимодействий между нуклеотидами в упрощенном случае можно считать невозможными (они возможны в виде исключения при некоторых редких условиях, но для нас это не важно). Уотсон-Криковское спаривание оснований еще называется комплементарностью.
В 1953 году Уотсон и Крик опубликовали свою модель структуры ДНК, оказавшейся абсолютно верной. ДНК (дезоксирибонуклеиновая кислота) — это интересно устроенный линейный полимер. Одна нить ДНК состоит из монотонно повторяющегося сахаро-фосфатного остова (он асимметричен и имеет направление, различают 5' и 3' конец цепи), однако к каждому сахару (дезоксирибозе в случае ДНК) прикреплен один из четырех нуклеотидов (синоним слова нуклеотид — «основание») — аденин, либо тимин, либо цитозин, либо гуанин. Обычно их обозначают одной буквой — А, Т, Ц, Г. Таким образом, в ДНК есть только 4 типа мономеров, в отличие от 20 аминокислот в составе белка, что делает структуру ДНК намного проще. Дальше становится еще веселей — есть так называемое «Уотсон-Криковское спаривание оснований»: аденин может специфично связываться с тимином, а гуанин — с цитозином, образуя пары А-Т и Г-Ц (и еще Т-А и Ц-Г, разумеется), другие взаимодействий между нуклеотидами в упрощенном случае можно считать невозможными (они возможны в виде исключения при некоторых редких условиях, но для нас это не важно). Уотсон-Криковское спаривание оснований еще называется комплементарностью. 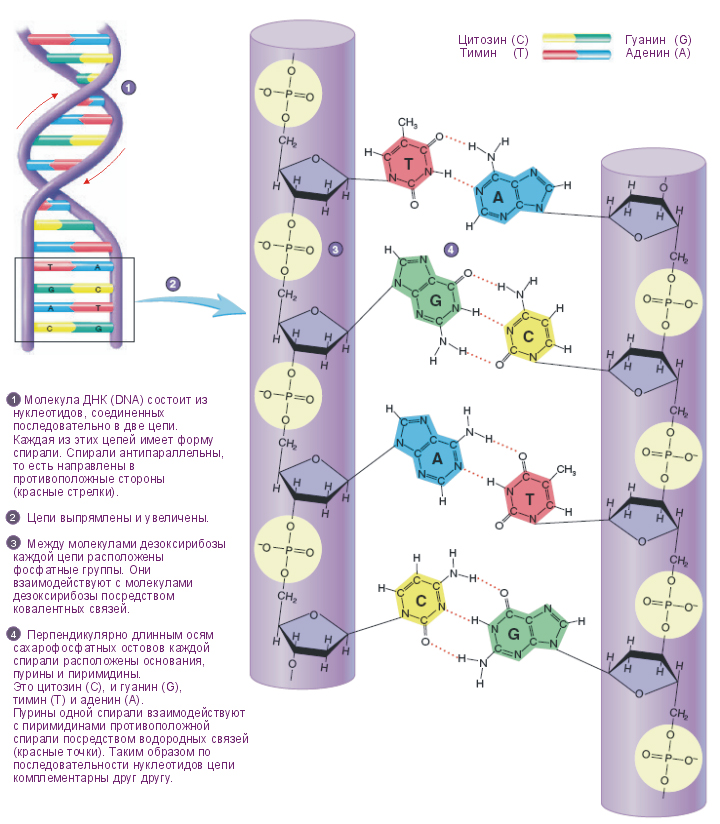
Две цепи ДНК, последовательность оснований которых комплементарна, немедленно «слипаются» в двойную спираль.Возникает вопрос: а что, если на одной цепи ДНК находятся две комплементарные области? Ответ: цепь ДНК может согнуться и комплементарные области смогут образовать двойную спираль, а вместе с местом изгиба эта структура будет называться «шпилькой» (DNA hairpin):

На чем же основано «слипание» двух комплементарных цепей ДНК (или, аналогично, двух комплементарных участков одной цепи)? Это взаимодействие держится на водородных связях. Пара А-Т соединяется двумя водородными связями, пара Г-Ц — тремя, поэтому эта пара более энергетически устойчива. Про водородные связи надо понимать следующее: энергия одной водородной связи (5 ккал/моль) не намного превосходит энергию теплового движения, а значит, одна отдельно взятая водородная связь может быть с высокой вероятностью тепловым движением разрушена. Однако, чем больше водородных связей, тем более устойчивой становится система. Это значит, что короткие участки комплементарных оснований ДНК не могут образовать устойчивую двойную спираль, она будет легко «плавиться», однако более длинные комплементарные участки уже смогут образовать стабильные структуры. Стабильность двухцепочечной структуры выражается одним параметром — температурой плавления (Тм, melting temperature). По определению, температура плавления — это температура, при которой в равновесии 50% молекул ДНК с данной длиной и последовательностью нуклеотидов находятся в двухцепочечном состоянии, а другие 50% — в расплавленном одноцепочечном состоянии. Очевидно, что температура плавления напрямую зависит от длины комплементарной области (чем длиннее — тем выше температура плавления) и от нуклеотидного состава (так как в паре Г-Ц три водородные связи, а в паре А-Т — две, то чем больше пар Г-Ц, тем выше температура плавления). Температура плавления для данной последовательности ДНК легко считается по эмпирически выведенной формуле.
От теории к практике
Итак, теорию мы изучили. Что же мы можем сделать на практике? С помощью химического синтеза мы можем напрямую синтезировать цепи ДНК длиной до 120 нуклеотидов (просто потом выход продукта резко падает). Если же нам нужна более длинная цепь, то ее без проблем можно собрать из тех самых химически синтезированных фрагментов длиной до 120 нуклеотидов (например, дядюшка Крейг Вентер отличился тем, что из кусочков собрал ДНК длиной аж 1,08 миллиона пар оснований). То есть в 21 веке мы можем легко и дешево делать ДНК любой последовательности, какой только захотим. А хотим мы, чтобы потом ДНК сворачивалась во всякие хитрые и сложные структуры, которые мы потом сможем использовать. Для этого у нас есть принцип комплементарности — как только в последовательности ДНК появляются комплементарные зоны, они слипаются и образуют двухцепочечный участок. Очевидно, мы хотим делать структуры, стабильные при комнатной температуре, значит мы хотим рассчитать температуру плавления для данных участков и сделать ее достаточно большой. При этом на одной цепи ДНК мы можем делать много разных областей с разными последовательностями и слипаться будут только комплементарные. Так как комплементарных областей может быть несколько, в результате молекула может свернуться достаточно сложным образом! Как-то так, например:

Так же, помимо положительного дизайна (создание областей, способных образовывать нужную нам структуру), при разработке структур с самосборкой нельзя забывать и о негативном дизайне — нужно проверять получившуюся последовательность ДНК на потенциальное наличие паразитных взаимодействий (когда части созданных нами областей оказываются способными взаимодейстовать по-другому, образуя ненужные нам паразитные структуры) и от этих паразитных структур и взаимодействий избавляться, меняя нуклеотидную последовательность ДНК. Как получить простейшие структуры ДНК типа «шпильки» достаточно очевидно, но скучно и неинтересно. Можно ли из ДНК сделать что-то посложнее? Здесь уже без компьютерных вычислений не обойтись. Мы хотим некую структуру и теперь должны подобрать последовательность ДНК, которая в эту структуру свернется за счет взаимодействия комплементарных областей, но при этом в последовательности не должно быть паразитных взаимодействий, непредусмотренных нами комплементарных областей, образующих альтернативные структуры. Плюс структура должна отвечать другим критериям, например, иметь температуру плавления выше некоторой заданной величины. В результате имеем типичную оптимизационную задачу.
Двухмерные структуры из ДНК
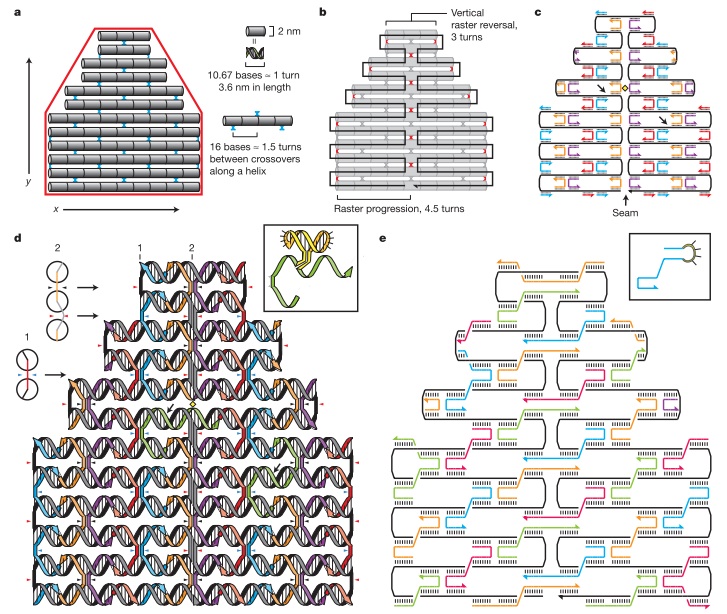
Методологический прорыв устроил Paul Rothemund (Калифорнийский Технологический Институт) в 2006 году, именно он и придумал термин «ДНК-оригами». В своей статье в «Nature» он представил множество забавных двухмерных объектов, сделанных из ДНК. Принцип, предложенный им, достаточно прост: взять длинную (примерно 7000 нуклеотидов )«опорную» одноцепочечную молекулу ДНК и затем с помощью сотни коротких ДНК-скрепок, образующих двухцепочечные области с опорной молекулой, согнуть опорную ДНК в нужную нам двухмерную структуру. Вот рисунок из оригинальной статьи, представляющий все стадии разработки. Для начала (а) нарисуем нужную нам форму красным цветом и прикинем, как заполнить ее ДНК (представим ее на этом этапе в виде труб). Далее (b) представим, как провести одну длинную опорную молекулу по нужной нам форме (показана черной линией). На третьем этапе (с) подумаем, где мы хотим разместить «скрепки», стабилизирующие укладку длинной опорной цепи. Четвертый этап (d): больше деталей, прикидываем, как будет выглядеть вся нужная нам структура ДНК и, наконец, (e) мы имеем схему нужной нам структуры, можно заказывать ДНК нужной последовательности!
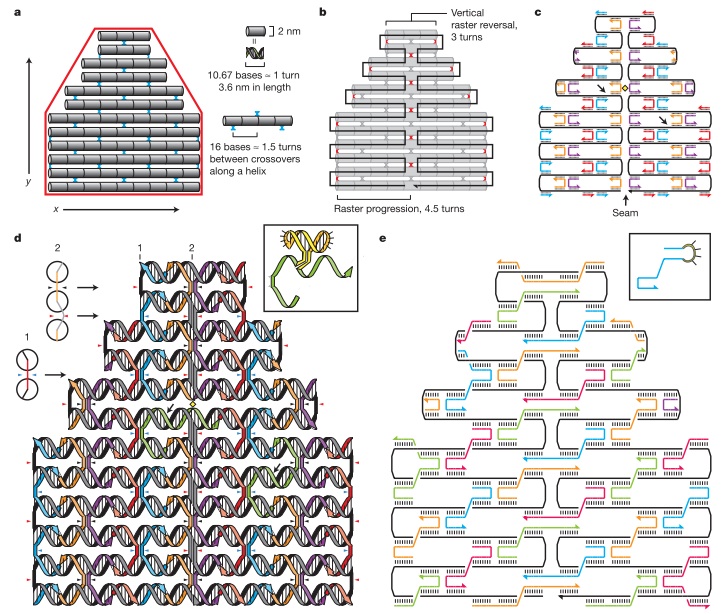
Как же из химически синтезированных ДНК собрать нужную нам структуру? Здесь на помощь приходит процесс плавления. Мы берем пробирку с водным раствором, бросаем в нее все фрагменты ДНК и нагреваем до 94-98С, температуры, которая гарантировано плавит всю ДНК (переводит ее в одноцепочечную форму). Далее мы просто очень медленно (в течении многих часов, в некоторых работах — в течении нескольких дней) охлаждаем пробирку до комнатной температуры (эта процедура называется «отжиг», annealing). При этом медленном охлаждении, когда температура оказывается достаточно низкой, постепенно образуются нужные нам двухцепочечные структуры. В оригинальной работе в каждом эксперименте примерно 70% молекул успешно собирались в нужную структуру, остальные имели дефекты.
Далее, после того, как структура рассчитана, неплохо бы доказать, что она собирается именно так, как нам надо. Для этого чаще всего используют атомно-силовую микроскопию, которая как раз прекрасно показывает общую форму молекул, но иногда используют и cryo-EM (электронную микроскопию). Автор сделал множество веселых форм из ДНК, на картинках представлены расчетные структуры и результат экспериментального определения структур с помощью атомно-силовой микроскопии. Наслаждайтесь!


Трехмерные структуры из ДНК
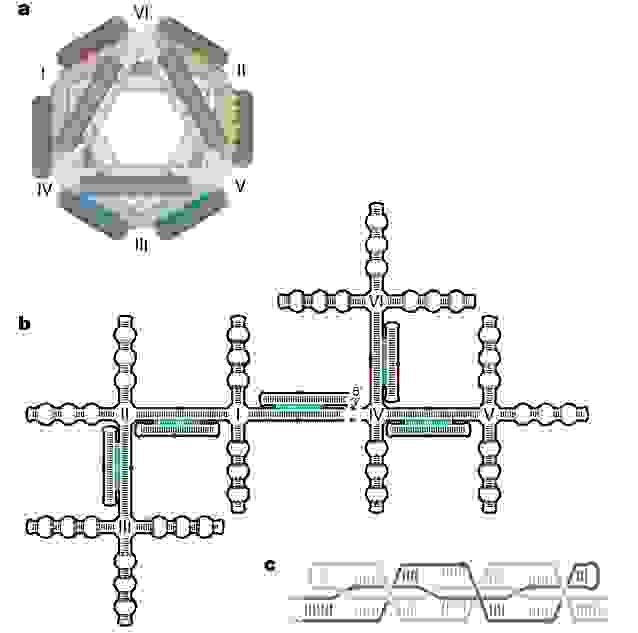
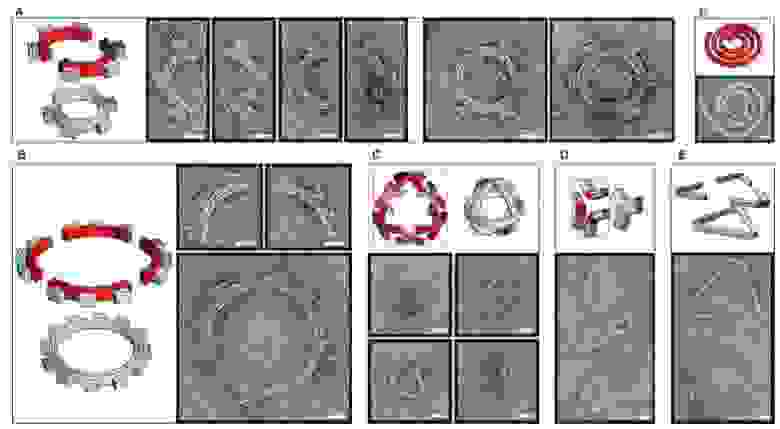
После того, как разобрались с конструированием сложных плоских объектов, почему бы не перейти к третьему измерению? Здесь пионерами была группа ребят из Института Скриппса в Ла-Холле, Калифорния, которые в 2004 году придумали, как из ДНК сделать нано-октаэдр. Хотя эта работа и сделана на 2 года раньше плоского ДНК-оригами, в тот раз был решен лишь частный случай (получение октаэдра из ДНК), а в работе по ДНК-оригами было предложено общее решение, поэтому именно работа 2006 года по ДНК-оригами считается основополагающей.
Октаэдр был сделан из одноцепочечной молекулы ДНК длиной примерно 1700 нуклеотидов, имеющей комплементарные области и к тому же скрепленой пятью 40-нуклеотидными ДНК-адаптерами, в результате был получен октаэдр с диаметром 22 нанометра.
На рисунке обратите внимание на цветовую кодировку на двухмерной развертке октаэдра. Видите области, отмеченные одинаковым цветом? Они содержат как комплементарные зоны (параллельные участки соединенные поперечными связями), так и некомплементарные (на схеме они изображены в виде пузырьков), при этом зоны одного цвета, расположенные в разных частях двухмерной развертки, взаимодействуют друг с другом, образуя сложную структуру, изображенную на рисунке 1с и образующую грань трехмерного тетраэдра. Наслаждайтесь красивыми картинками!

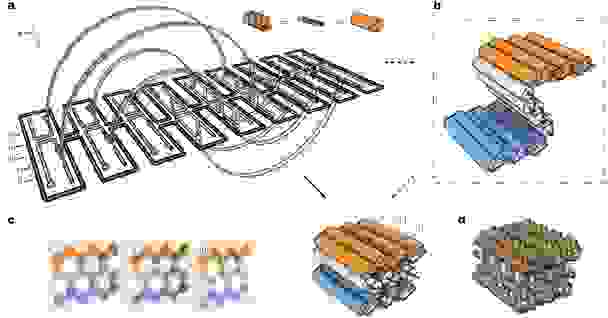
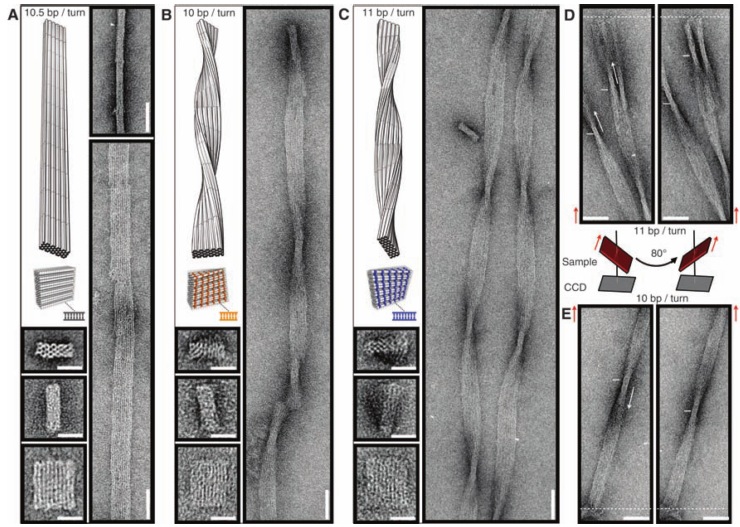
В 2009 году ученые из Бостона и Гарвардского Университета опубликовали принципы построения трехмерного ДНК-оригами, как они сами говорят, по подобию пчелиных сот. Одно из достижений этой работы — люди написали open-source программу caDNAno для конструирования трехмерных структур ДНК (она работает на Autodesk Maya). С этой программой даже неспециалист может собрать нужную структуру из готовых блоков с использованием простенького графического интерфейса, а программа рассчитает необходимую последовательность (или последовательности) ДНК, в эту структуру сворачивающуюся.



В следующей своей работе они научились делать из ДНК сложные трехмерные объекты с контролируемым искривлением и порадовали читателей журнала «Science» красивыми картинками разных искривленных объектов из ДНК (там такие классные шестеренки получились!).



Периодические структуры
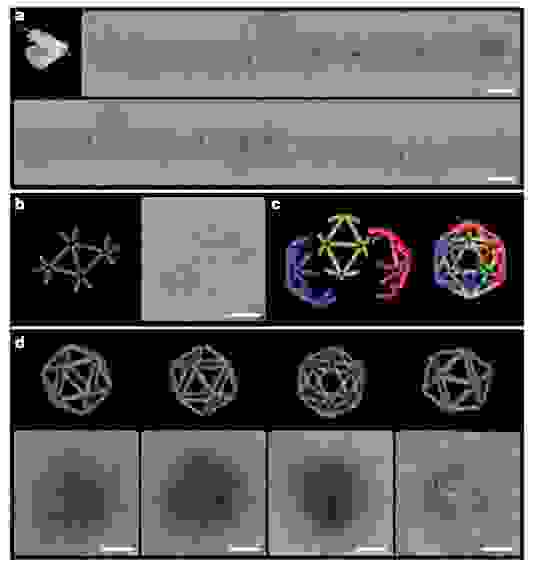
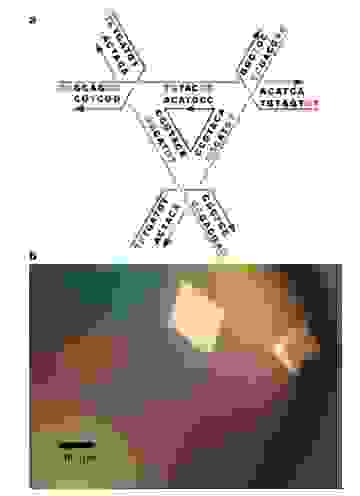
 До сих пор ученые игрались с непериодическими структурами из ДНК. А что если сделать такую структуру, чтобы один блок мог взаимодействовать с другим таким же блоком, и так до бесконечности? Представьте себе три отрезка, расположенных под углом 90 градусов друг к другу (походит на противотанковый еж). Очевидно, что такая структура может быть узлом бесконечной кубической решетки, если каждая сторона такого ежа будет взаимодействовать с другим таким ежом. Именно эту идею в 2010 году воплотили на практике ученые из Нью-Йорка, они сделали такого ДНК-ежа, который немедленно сформировал трехмерную решетку, то есть кристалл из ДНК, так что они использовали рентгеноструктурный анализ, чтобы показать, что ДНК образовали именно такую структуру, какую они и хотели. В свою очередь, так как кристаллы ДНК имели размер до пол-миллиметра (а это уже макро-объект), было гордо заявлено, что теперь из нано-объектов мы умеем собирать макро-объекты.
До сих пор ученые игрались с непериодическими структурами из ДНК. А что если сделать такую структуру, чтобы один блок мог взаимодействовать с другим таким же блоком, и так до бесконечности? Представьте себе три отрезка, расположенных под углом 90 градусов друг к другу (походит на противотанковый еж). Очевидно, что такая структура может быть узлом бесконечной кубической решетки, если каждая сторона такого ежа будет взаимодействовать с другим таким ежом. Именно эту идею в 2010 году воплотили на практике ученые из Нью-Йорка, они сделали такого ДНК-ежа, который немедленно сформировал трехмерную решетку, то есть кристалл из ДНК, так что они использовали рентгеноструктурный анализ, чтобы показать, что ДНК образовали именно такую структуру, какую они и хотели. В свою очередь, так как кристаллы ДНК имели размер до пол-миллиметра (а это уже макро-объект), было гордо заявлено, что теперь из нано-объектов мы умеем собирать макро-объекты.
Вот стерео-картинка узла решетки, если умеете правильно скашивать глаза (это прямая стереопара), можете посмотреть в 3D (на нижней картинке с электронной плотностью ДНК четко видны два узла-«противотанковых ежа»):

Динамические структуры
Следующий шаг в конструировании трехмерных нано-объектов так же очевиден — а что если заставить это все как-то двигаться? Движущийся объект уже можно и нано-роботом назвать. Самый простенький ДНК-шагоход был сделан в 2008 году командой из Калифорнийского Технологического института. Работает он по достаточно простому принципу. Представьте себе одноцепочечную ДНК длиной, скажем, 100 нуклеотидов. Представьте другую одноцепочечную ДНК, короткую, длиной 50 нуклеотидов, комплементарную половине первой молекулы. Что будет, если их смешать? Правильно, они образуют двухцепочечную структуру в районе этих 50 нуклеотидов, вторая же половина первой молекулы останется свободной. А что если к этой структуре добавить еще одну молекулу ДНК, длиной 100 нуклеотидов и полностью комплементарной первой молекуле? Ответ вполне очевиден: она вытеснит короткую цепь длиной 50 нуклеотидов, так как обладает большим сродством к первой молекуле (у них сродство 100 из 100, а с короткой — только 50 из 100 нуклеотидов). Именно так и работал первый ДНК-шагоход. На подложке закреплены молекулы одноцепочечной ДНК, к ним из раствора приходит молекула-шагоход, имеющая комплементарные зоны к двум соседним цепям на подложке и связывается с ними. Если потом мы добавим в раствор другую ДНК, имеющую большее сродство к первой цепи на подложке, то она вытеснит одну ногу шагохода, после чего эта нога свяжется со следующей (третьей) цепью ДНК на подложке. Добавляя новые вытесняющие ДНК можно гнать шагоход все дальше и дальше по подложке. Обратный ход невозможен, так как предыдущие цепи на подложке уже инактивированы связыванием с более длинной молекулой ДНК.
Хотя первый шагоход выглядел настолько примитивно, конструкция была доработана учеными из Нью-Йорка. Они сделали более сложный шагоход с несколькими «руками» и «ногами», прицепили при помощи комплементарных ДНК на подложку «груз» (золотые частицы диаметром 5 и 10 нм) и «запрограммировали» шагоход таким образом, чтобы он прошел по подложке и собрал груз — три маленькие и одну большую золотые частицы. Последовательность шагов легко отследить по стрелочкам, а на экспериментальной картинке справа видны частицы золота и как шагоход их собирает. Нано-робот в действии! На нижней картинке показано, как именно происходит процесс «шагания» и сбора груза, принцип — тот же самый, вытеснение одной ДНК другой.

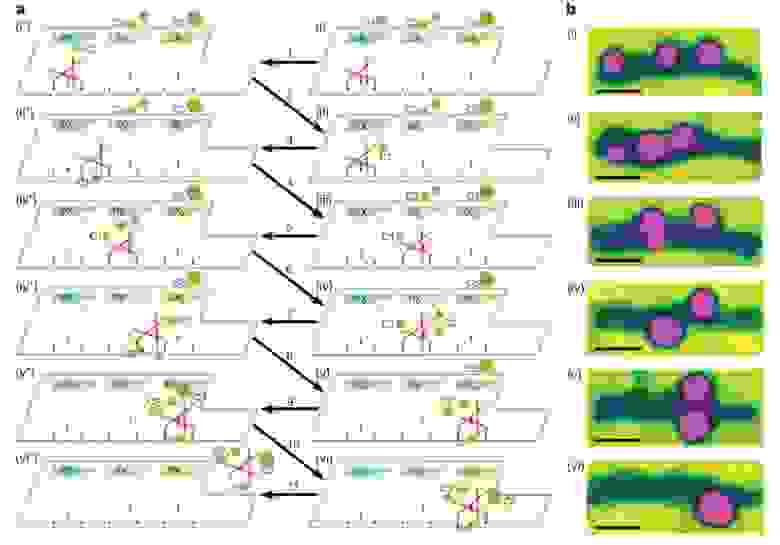
Но это еще не все. Венцом ДНК-робототехники (думаю, тут в моем голосе слышим некоторый сарказм) стал «наноробот для молекулярного транспорта», как окрестили его создатели из Бостона. Фактически ребята сделали некую «коробочку» из ДНК, закрывающуюся на «замок» из ДНК, который может быть открыт по уже известному нам принципу вытеснения одной ДНК другой, как в шагоходах. Внутри коробочки спрятан груз — частицы золота либо молекулы иммуноглобулина. ДНК можно химически модифицировать таким образом, чтобы на ней можно было закрепить этот самый груз. Итак, мы имеем закрытую коробочку, содержимое коробочки надежно спрятано. Тут мы добавляем молекулу, открывающую коробочку, она открывается и иммуноглобулин, спрятанный внутри, выходит наружу и начинает действовать! Мы же хлопаем в ладоши, умиляемся и радуемся прогрессу.

Ребята даже не поленились сделать демонстрацию proof of principle на живых раковых клетках: они прятали в коробочку антитела, блокирующие ключевые белки клеточного цикла, вводили коробочки в раковые клетки и после добавления открывающего коробочку активатора раковые клетки правда переставали делиться! Таким образом была показана принципиальная возможность использования таких конструкций для направленной доставки лекарств в организме и их выделения в нужное время по сигналу от молекулы-активатора. Одна осталась проблема, как раковую клетку от здоровой надежно отличить…
Для чего козе баян?
Все это, конечно, здорово и хорошо, картинки красивые, но внимательный читатель может спросить: «А где обещанная в самом начале польза для народного хозяйства?». Как и со всякой новой технологией, пока большой практической пользы действительно нет, помимо эстетического удовольствия от созерцания этой нано-красоты. Однако, все только начинается! Во-первых, ДНК может быть химически модифицирована, к ней могут быть добавлены химические группы, обеспечивающие связывание с другими молекулами и тогда ДНК можно использовать как подложку для построения сложных структур из других молекул. Например, сейчас все хотят что-то мастерить из нанотрубок. Если удастся сделать адаптер, связывающийся одним концом с ДНК, а другим — с нанотрубкой, тогда структуры из ДНК можно использовать для соединения нанотрубок. С другой стороны, уже есть сообщения о контролируемой металлизации ДНК, а отсюда уже рукой подать до конструирования электронных устройств на базе структур из металлизированной ДНК. Может быть, ученые изобретут более подходящий полимер, обеспечивающий более удобную самосборку сложных структур, но в любом случае ДНК-оригами займет свое место в истории науки, как один из первых примеров конструирования сложных объектов в нано-масштабе. Как бы то ни было, нас ждет большое и светлое будущее, чего и вам желаю!
PS: интересное дополнение от vxsw:
«Уже пару лет проводится конкурс BIOMOD по дизайну таких штук при поддержке Wyss Institute (замеченного во многих интересных биотехнологических достижениях вроде недавней 3D-печати мини-аккумуляторов).»
PPS: в личке спросили, почему ДНК, а не РНК. Ответ такой: я вижу две основные причины: (1) ДНК — химически более стабильна. Все живые организмы синтезируют огромное количества РНКаз, ферментов, уничтожающих РНК. Если Вы случайно залезете голым пальцем в пробирку с РНК, от РНК ничего не останется — все сожрут РНКазы. Поэтому с РНК работают в специальных помещениях и тд — мороки гораздо больше, чем при работе с ДНК. С ДНК таких проблем нет, палец в пробирку сунешь — ничего ДНК не будет. (2) Стоимость химического синтеза РНК в разы превышает стоимость синтеза ДНК. Думаю, поэтому народ и развлекается с ДНК — дешевле и проще.











